爱必信(上海)生物科技有限公司品牌商
14 年
手机商铺
- NaN
- 0
- 0
- 2
- 2
推荐产品
公司新闻/正文
mRNA 质量检测之 poly A 尾检测方法
2057 人阅读发布时间:2023-04-04 10:48
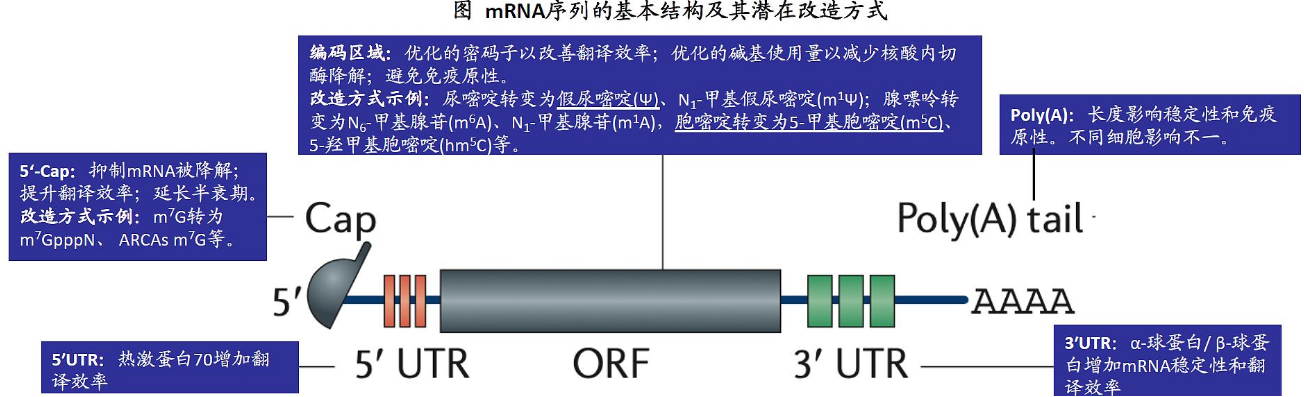
首先我们需要先回顾下 3’端 polyA 尾的功能:保护帽结构不被降解,与 polyA 结合蛋白、5’ Cap(帽结构)和翻译起始因子蛋白协同作用,启动蛋白质的翻译。多数 mRNA 的 polyA 长度为 50–200 nt,以维持正常的生物学功能。polyA 尾的有无与长度是 mRNA 药物的关键质量属性之一。
当前 polyA 尾的检测分为两种思路:
① 与加帽率的分析策略类似,酶切后纯化回收 3’端 polyA 尾,随后通过毛细管电泳(CE)、变性聚丙烯酰胺凝胶电泳(PAGE)、高效液相色谱(HPLC)或液相色谱质谱联用(LC-MS),对 polyA 尾进行定性和定量分析。
② 基于二代或三代测序技术表征 polyA,已报道的测序方法包括 TAIL-seq、PAL-seq、FLAM-Seq 和 PAIso-Seq 等。
2018 年诺华公司 Beverly M 等发表在 Anal Bioanal Chem 杂志的一篇文章,《Poly A tail length analysis of in vitro transcribed mRNA by LC-MS》,研究基于 RNAse T1 酶切与 oligo dT 磁珠纯化得到 polyA 尾,使用液相色谱质谱联用(LC-MS)检测 polyA 尾长度的分布,目前该策略是当前 mRNA 体外制备工艺中较为常用的方法。
2022 年 Brouze A 等发表在 Wiley Interdiscip Rev RNA 期刊的一篇综述,《Measuring the tail: Methods for poly(A) tail profiling》,总结了利用二代(Illumina)或三代(PacBio)测序与 RNA 直接测序平台检测 polyA 尾,可在分析 polyA 长度分布的同时表征序列的完整性和准确性。
01 基于内切酶与 LC-MS 平台的 polyA 尾检测
1)mRNA 样品制备
一步法加尾:设计模板序列,包括不同长度 polyA 尾(27-112 nt),体外转录(IVT)制备 mRNA 样品。
酶法加尾:模板序列不包括 polyA,使用大肠杆菌来源的 polyA 聚合酶进行 mRNA 的加尾修饰。制备得到的 mRNA 进行琼脂糖凝胶电泳,确认 mRNA 样品的长度和完整性。以 10 nt 和 20 nt 的 polyA 为标准品。
2)RNAse T1 酶切与磁珠纯化
① 酶切:内切酶 RNAse T1 可特异性地在单链 RNA 的鸟嘌呤核糖核苷酸(G)后进行切割。100 pmol mRNA 加热至 95℃ 后迅速降至 25℃,随后加入 10×RNase H 缓冲液、2 μl RNase T1 酶(100 μl 体系),37℃ 孵育 3 小时。
② 纯化:通过 polyA 尾与 oligo dT 磁珠特异性结合进行纯化,使用 0.1 M 醋酸铵溶液清洗磁珠,随后加入 75% 甲醇,80℃ 孵育 1 min,洗脱 polyA 片段。
③ 换液:通过旋转蒸发去除甲醇,重悬于含 1% 甲醇的 0.1 mM EDTA 溶液,用于 LC-MS 分析。
3)LC-MS 分析
使用高分辨液相质谱联用仪,固定相为 ACQUITY C18 色谱柱,流动相 A 为 200 mM 六氟异丙醇+8.15 mM triethylamine(pH 7.9),流动相 B 为100%MeOH。
洗脱条件为:5%B 洗脱 1 min,1-12 min 内 B 浓度由 5% 线性增加到 25%,然后 90%B 冲洗 1min 后恢复 5%B。紫外波长设置为 260 nm。
4)不同加尾方法得到的 polyA 尾长度评估
与利用质粒模板转录生成 polyA 相比,酶法加尾的 mRNA 样品的出峰更宽,polyA 尾长度的分散性更大。
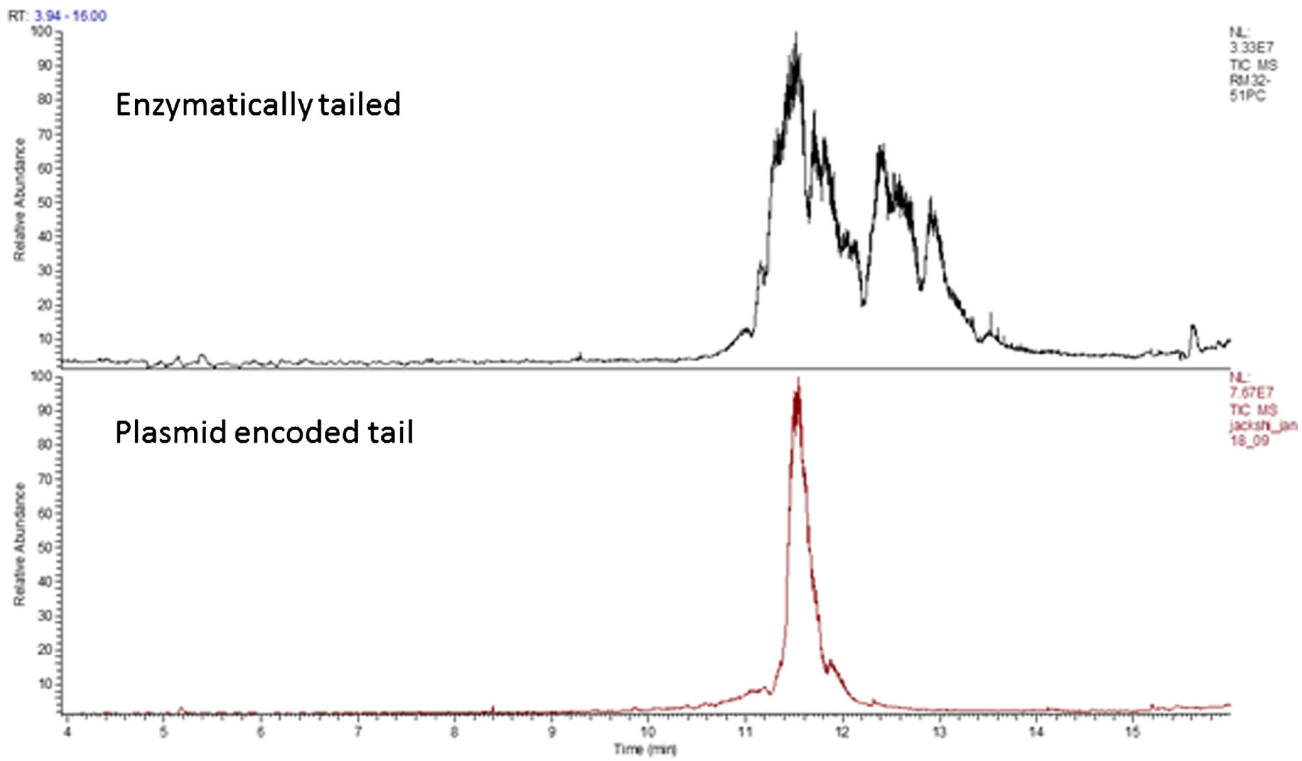
图 1 不同加尾方法得到的 polyA 尾分布(离子色谱图)
5)HPLC 与 MS 的分辨率
当 polyA 长度为 27 nt 时,高效液相色谱(HPLC)可有效分离相差一个腺苷的 polyA,但当碱基个数增加至 64 或 100,HPLC 未能有效分离相差一个腺苷的 polyA(图 2a)。而质谱(MS)可弥补这一缺陷,当 polyA 长度为 100 nt,电喷雾质谱图可精确检测任一不同分子量的 polyA(即不同长度的 polyA),通过峰强度对 polyA 长度分布进行相对定量(图 2b)。
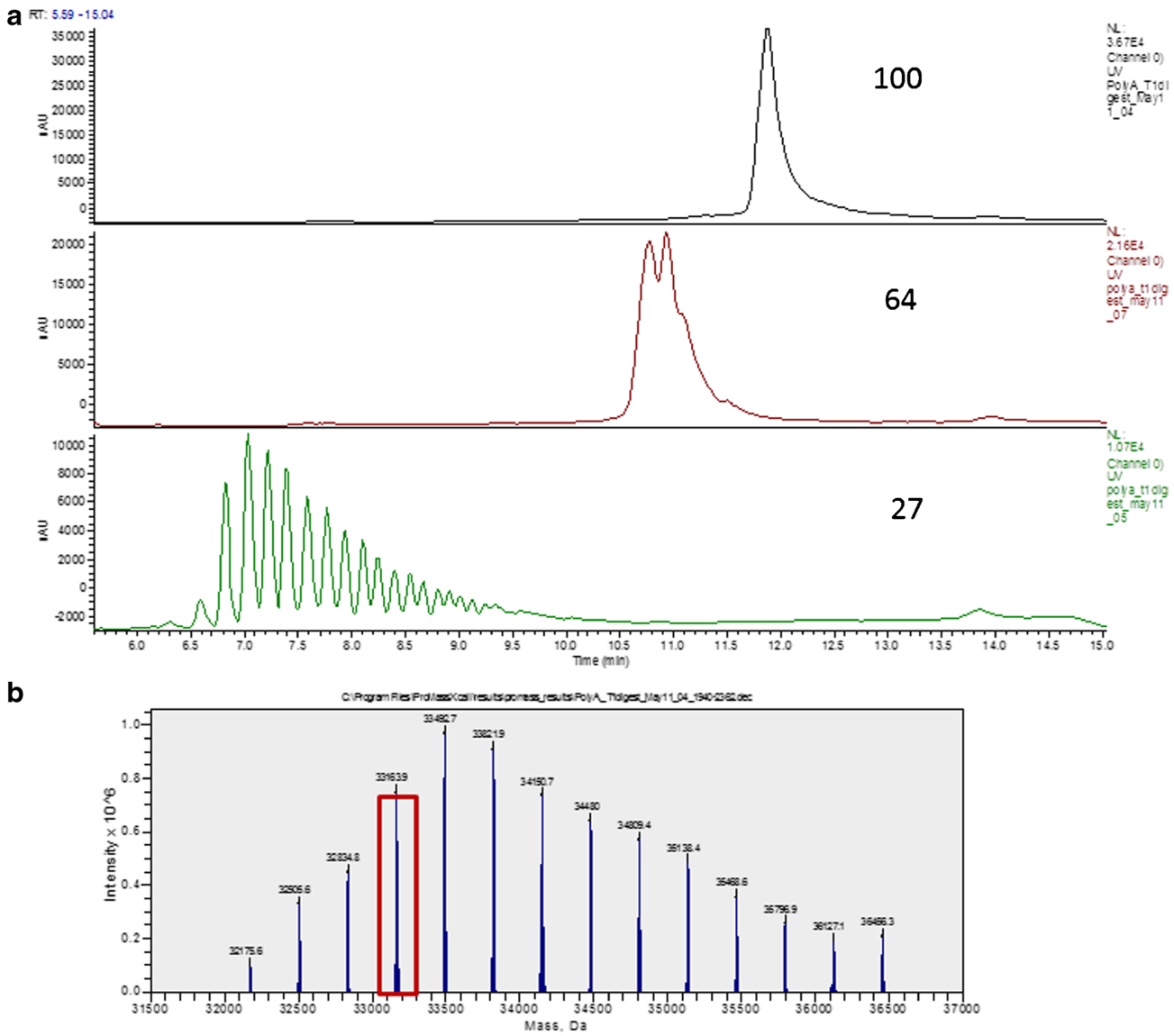
如上所述,文章介绍了一种分析 mRNA polyA 尾长的方法。该方法基于 RNase T1 将 polyA从mRNA 序列上切割下来,通过 dT 磁珠特异性捕获 polyA,然后借助 LC-MS 的高分辨率,定量分析 mRNA 的 polyA 尾长度分布。
基于该策略的可替代方法包括:根据核苷酸序列特性,使用其他 RNA 酶(如 RNase A 或 RNase H)代替 RNase T1,以获得 polyA 片段,随后通过毛细管电泳、HPLC 对短核苷酸进行分离与定量分析。
02 基于测序平台的 polyA 尾检测
有研究报道,可利用 Illumina 二代测序、PacBio 三代测序与纳米孔 RNA 直接测序平台分析 polyA,测序原理及流程如下:
1)基于 Illumina 平台分析 polyA 尾
Illumina RNA-Seq 的原理是通过富集 mRNA(polyA 富集)或去除 rRNA,产生片段化的 RNA 序列,然后将 RNA 逆转录生成双链 cDNA,在片段末尾加入测序接头后进行 PCR 扩增。cDNA 分子聚集在流动池,通过 DNA 合成体系中的 3’端封锁荧光标记的核苷酸底物读取荧光信号,从一端(单端测序)或两端(成对末端测序)进行测序。
常用的方法包括 TAIL-Seq 和 PAL-Seq,借助于制备完整的包括 polyA 尾在内的 3’端 mRNA 测序文库。首先将 RNA 与生物素化的 3’端 DNA 接头连接,并用 RNase T1 部分酶切,留下完整的 polyA 尾,随后使用链霉亲和素磁珠捕获 3’端片段。通过凝胶电泳筛选不同条带,并与 5’端接头连接,为逆转录、文库扩增和测序提供完整的模板。如图 3 所示,PAL-Seq 和 TAIL-Seq 的不同在于 DNA 接头,TAIL-Seq 仅存在一个单链 DNA 接头,测序过程需去除 rRNA。而 PAL-Seq 技术除单链 DNA 接头外,还存在一端与 polyA 尾和 3’端接头互补的 DNA 寡核苷酸序列,提高了连接效率,因此不需去除 rRNA。
Illumina 测序技术的不足在于,由于 polyA 的长多聚物特性,容易产生 PCR 扩增的偏倚,保真度较低,序列准确性较低。
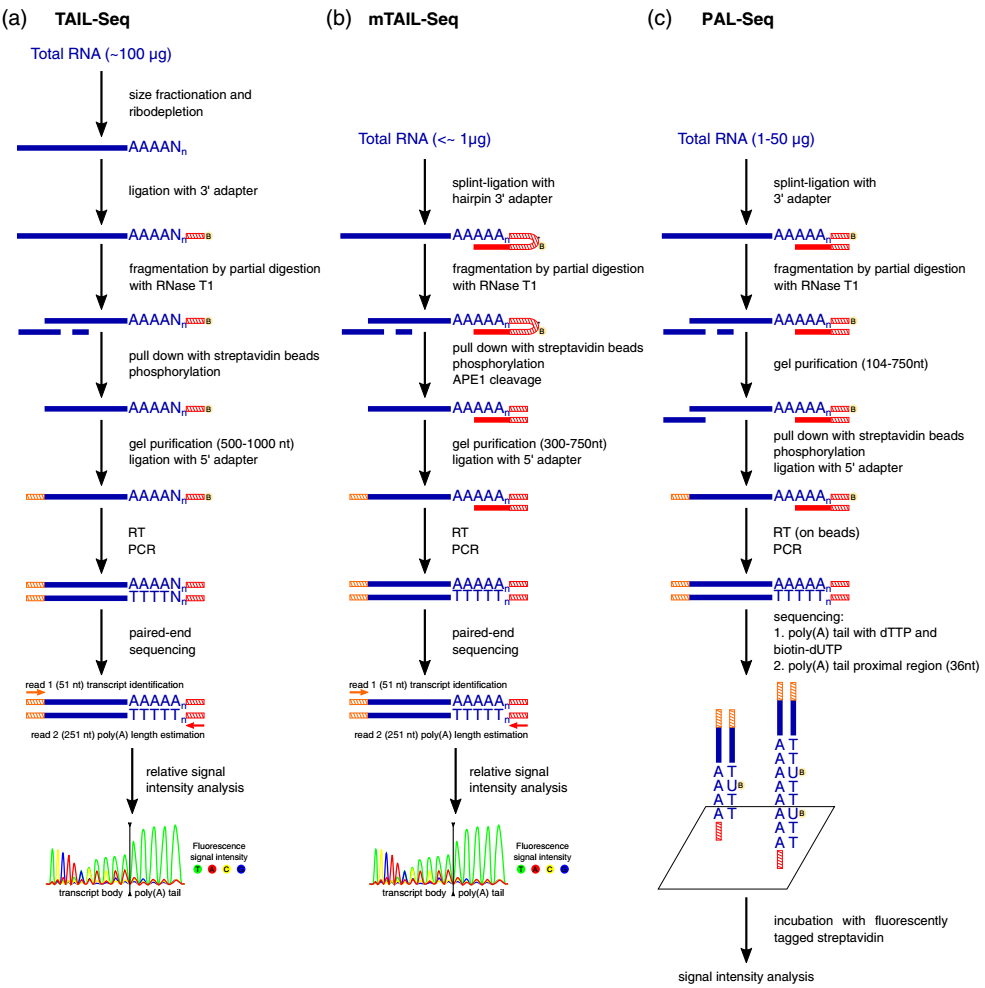
图 3 基于 Illumina 平台的 polyA 尾测序方法
2)基于 PacBio 平台分析 PolyA 尾
不同于 Illumina 短 cDNA 片段合成测序的方法,PacBio 采用单分子实时测序技术,在处理均聚物的效果优于 Illumina,可对全长 RNA 分子进行测序,提供有关 polyA 的长度和序列信息。
常用方法包括 FLAM-Seq 和 PAIso-Seq。如图 4所示,两种方案的主要步骤是 3’端延伸、逆转录、cDNA 扩增、环状接头连接和 PacBio 测序。
二者的不同点为:
FLAM-Seq 首先选择带有 polyA 尾的 mRNA,随后对其进行 G/I 加尾。逆转录引物由 5’末端的 PCR handle 组成,然后在模板置换寡核苷酸(TSO)的情况下合成第二链,最后将合成的双链 cDNA 扩增并进行 PacBio 测序。而 PAIso-Seq 首先与 oligo dT 结合,3’末端延伸,经 USER 降解 oligo dT 寡核苷酸,随后经逆转录、PCR 扩增双链 cDNA 并进行 PacBio 测序。PacBio 具备均聚物测序能力,可用于分析 polyA 尾的序列组成。
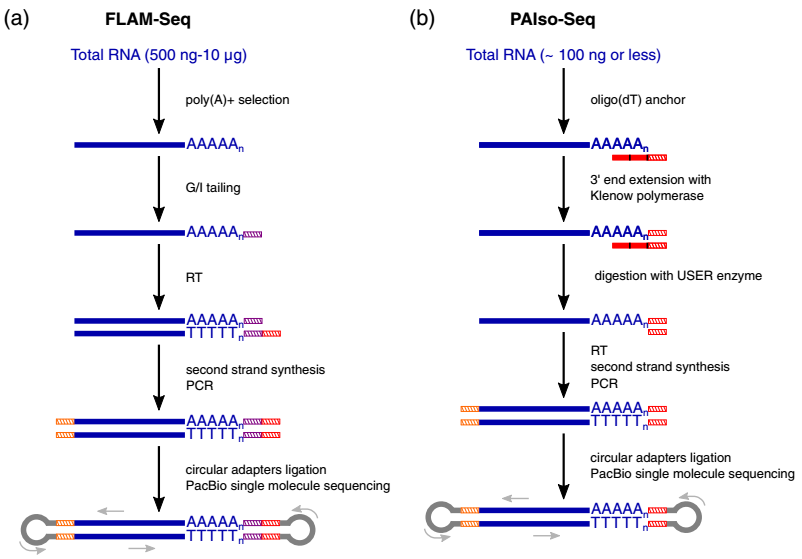
图 4 基于 PacBio 平台的 polyA 尾测序方法
3)基于纳米孔 RNA 直接测序平台分析 polyA 尾
纳米孔 RNA 直接测序平台由 Oxford NanoporeTechnologies(ONT)推出,与二代、三代测序相比,纳米孔 RNA 直接测序技术分析 polyA 尾不需进行 PCR,流程如图 5 所示,将 3’端接头连接至 mRNA(含 polyA),逆转录(可选操作)后连接至3’端测序接头(与动力蛋白连接),随后被加载到纳米孔形成的流动池,施加电压后,在动力蛋白驱动下,RNA 由 3’端至 5’端通过纳米孔,根据特异性电流信号分辨碱基序列。纳米孔 RNA 直接测序平台不需切断 mRNA 序列,直接读取全长序列;该平台不依赖于逆转录和 PCR 反应,可以同时表征序列的完整性、准确性和核苷酸修饰。
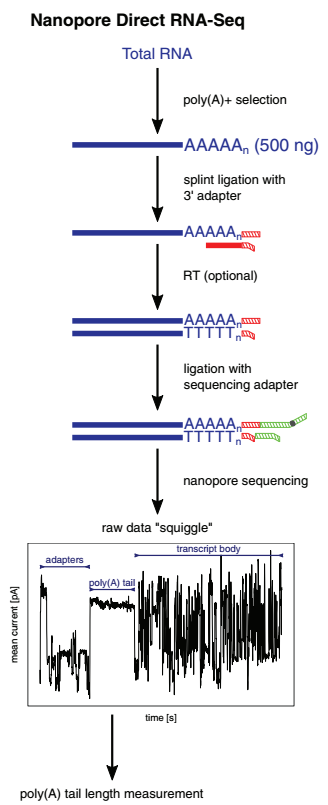
图 5 基于纳米孔 RNA 直接测序平台的 polyA 尾检测方法
03 结语
2018 年 Beverly M 等使用内切酶 RNase T1 进行特异性切割,利用磁珠分离得到 5’端 polyA 尾,随后通过 LC-MS 有效分离不同分子量大小的核苷酸序列,从而定量分析 polyA 尾长度分布。该方法是体外合成 mRNA polyA 尾的常用表征方法。
随着测序技术的发展,科学家们开始基于测序平台分析 polyA 尾长度。基于 Illumina 平台的 TAIL-Seq 和 PAL-Seq 技术,以及第三代测序技术 PacBio 平台,通过构建 cDNA 文库对 polyA 尾或全长 mRNA 进行测序,可检测 polyA 尾长度和序列完整性。但二者均依赖于 PCR 扩增 cDNA,长均聚物的存在导致 PCR 扩增不可避免地存在偏差,可能对 polyA 尾长和序列准确性造成影响。ONT 开发的直接测序方法,不依赖于逆转录与 PCR 扩增,可以实现 polyA 尾部长度及序列准确性的检测,不存在 PCR 偏倚性。然而以 TAILseq、PALseq、FLAMseq、PATseq 及纳米孔测序为代表的测序技术通量大、设备要求高,鉴于体外合成的 mRNA 通量较小,无法体现测序技术的高通量优势。
参考文献:
[1] Beverly M, Hagen C, Slack O. Poly A tail length analysis of in vitro transcribed mRNA by LC-MS. Anal Bioanal Chem. 2018 Feb;410(6):1667-1677. doi: 10.1007/s00216-017-0840-6.
[2] Brouze A, Krawczyk PS, Dziembowski A, et al. Measuring the tail: Methods for poly(A) tail profiling. Wiley Interdiscip Rev RNA. 2022 May 26:e1737. doi: 10.1002/wrna.1737.
[3] 药品审评中心. 新型冠状病毒预防用mRNA疫苗药学研究技术指导原则(试行). 2020.
[4] USP. Analytical Procedures for mRNA Vaccines–Draft. 2022.
转自耀海生物
逆转录系列产品推荐
| 货号 |
产品名称 |
规格 |
| Reverse Transcriptase II |
10000U/10000U×5 |
|
| Plus All-in-one 1st Strand cDNA Synthesis SuperMix (gDNA Purge) |
50T/100T |
|
| First-strand cDNA Synthesis Mix With gDNA Remover |
50T/200T |
|
| 逆转录一管化三代预混液 |
100T |
|
| One-Step RT-PCR Kit |
200T |
Absin特色产品线: